群体遗传学习笔记-基础知识
群体遗传学介绍
传统群体遗传学是基于观察到的等位基因频率与预期频率的分析。例如,在Wright-Fisher模型下,你可能会看到有性繁殖的二倍体个体群体,而且这些种群没有重叠世代。该模型忽略了诸如突变,重组,选择或种群大小或结构变化等影响。更复杂的模型可以包含在实际群体中观察到的影响的不同方面。然而,大多数这些模型都假设群体是以性方式繁殖的。
群体是指生活在一定空间范围内,能够相互交配并生育具有正常生殖能力后代的同种个体群。群体与个体相对,是个体的共同体,不同个体按某种特征结合在一起,进行共同活动、相互交往,就形成了群体。
有效群体大小:指与实际群体有相同基因频率方差或相同杂合度衰减率的理想群体含量,通常小于绝对的群体大小。
A locus (基因座)是基因组中的一个位置,我们可以在不同的个体中观察一个或几个等位基因。假定群体遗传学中使用的基因座是选择性中性的,可以是anonymous或非编码区,如微卫星基因座(SSR),单核苷酸多态性(SNP)。
A genotytpe (基因型)是特定基因座上给定个体携带的等位基因的组合。携带同一组等位基因的个体被认为具有相同的多基因座基因型 (MLG)。
基因型频率,群体中某一基因型个体占群体总个数的比例。可以反映某一基因型个体在群体中的相对数量。在群体遗传学中基因型频率指在一个种群中某种基因型的所占的百分比。
群体中基本度量标准是多态性,等位基因频率和基因型频率。多态性可以通过多种方式进行估计,例如观察到的多个等位基因的总数。
等位基因频率是群体遗传学的术语,用来显示一个种群中基因的多样性,或者说是基因库的丰富程度。在一个群体中,等位基因频率即某类等位基因占该基因位点上全部等位基因数的比率。如:在某种群中一个等位基因的基因频率为20%,那么在种群的所有成员中,1/5的染色体带有那个等位基因,而其他4/5的染色体带有该等位基因的其他对应变种—可以是一种也可以是很多种。
遗传平衡定律(哈迪-温伯格定律):是指在理想状态下,各等位基因的频率和等位基因的基因型频率在遗传中是稳定不变的,即保持着基因平衡。例:当等位基因只有一对(Aa)时,设基因A的频率为p,基因a的频率为q,则A+a=p+q=1,AA+Aa+aa=p2+2pq+q2=1。哈迪-温伯格平衡定律(Hardy-Weinberg equilibrium) 对于一个大且随机交配的种群,基因频率和基因型频率在没有迁移、突变和选择的条件下会保持不变。
群体分析
群体分析一般可以分为,分层分析和选择分析。分层分析可以定义为,基于群体内个体之间基因序列上的差异。群体选择分析是在群体水平在基因组不同区域遗传多样性的差异。
分层分析:
- 系统发育研究:一般使用发育树研究具有共同祖先之间进化关系,探究物种进化的关系和分类的关系。
- PCA(主成分分析):用较少不相关的变量替代原始大量相关的变量,来研究群体分层,亚种之前的进化关系。
- 群体结构分析:研究大群体中存在基因频率不同的亚群,一般可以用来推断祖先群,个体血缘组成,还有杂交事件。
选择分析
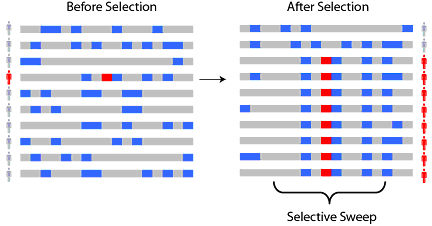
选择清除分析:自然选择促使有利突变在群体中保留下来,与之连锁的中性位点突变频率提升,非连锁的中性位点突变频率下降;简单的说就是基因组某区域由于受到了选择而消除多态性,即遗传多样性降低,在群体中出现高频的等位基因和低频的等位基因。主要用于:挖掘驯化过程中受选择的基因和挖掘物种适应性进化过程中受选择的基因。
适合度-分析是指生物体或生物群体对环境适应的量化特征,是分析估计生物所具有的各种特征的适应性,以及在进化过程中继续往后代传递的能力的指标。适合度是衡量一个个体存活和繁殖成功机会的尺度。适合度越大,个体成活的机会和繁殖成功的机会也越大,反之则相反(因此义项与广义适合度相对应,故亦可称之为狭义适合度)。
计算方式:适合度可以用数据计算出来:W=ml。其中,W代表适合度,m表示基因型个体生育力,l表示基因型个体存活率。
常用的统计方法:
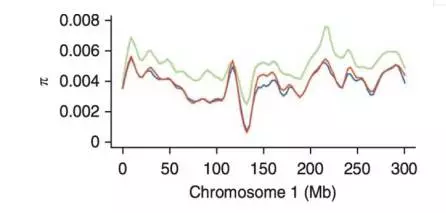
θπ、θW,Tajima's D,Fst。
θπ:群体中任意两条不同序列(个体)的碱基差异数(SNP)取平均值。
- 手人工选择的群体,遗传多样性相对单一,θπ值较小
- 野生群体遗传多样性大,θπ值比较大
- 单个群体内部基因型多样性(0-1),多样性越大,θπ越大。
θW:基于全部序列内分离位点个数
 此处输入图片的描述
此处输入图片的描述
中性检验(Tajima's D):
 此处输入图片的描述
此处输入图片的描述
Tajima’D = (θπ–θW)/Var(θπ–θW)
- θπ = θw Tajima's D = 0,中性进化;
- θπ < θw Tajima’s D < 0:群体中存在许多低频率的等位基因(稀有等位基因),则θW增大而θT不受影响(或影响较小),由定向选择或群体扩张引起;
- θπ > θw Tajima’s D > 0:群体中高等/中等频率的等位基因较多, θT增大而θW 不受影响,由于平衡选择和瓶颈效应引起的。
平衡选择与定向选择都属于正选择的范畴,因此,只要D值显著背离0,就可能是自然选择的结果;而当D值不显著背离0时,则中性突变。
Fst:群体间遗传分化指数,是种群分化和遗传距离的一种衡量方法,分化指数越大,差异越大。
Fst= (πBetween-πWithin)/πBetween
πBetween:来自群体间的所有两两个体间差异的均值
πWithin:来自亚群内所有两两个体间差异的均值
正选择vs负选择
 此处输入图片的描述
此处输入图片的描述
positive selection (正选择):自然选择“选留”一些稀少的等位基因,拥有这些等位基因的个体能繁殖更多的后代;这样的突变基因往往具有与原来基因不同的功能,而且该功能使得拥有它的生物更能适应环境。
negative selection(负选择):指群体中出现有害突变等位基因时,携带该等位基因的个体会因为生存力或育性降低而从群体中淘汰,也叫净化选择。
选择清除vs背景选择
 此处输入图片的描述
此处输入图片的描述
选择清除:在有利突变产生后被正选择固定的过程中,与之连锁的中性位点的变异也被固定。
背景选择:负选择在清除有害突变时,也会随之清除与其连锁的中性位点的变异。
选择清除和背景选择都会导致基因组上受选择的区域遗传多样性下降,两者很难区分,但背景选择在群体中不会导致高频等位基因突变出现。
连锁不平衡(Linkage disequilibrium, LD):指群体内不同位点等位基因间的非随机性组合的关系,即当位于同一条染色体的两个等位基因(A,B)同时存在的概率,大于群体中因随机分布而同时出现的概率时,就称这两个点处于LD状态。通常用D’和r2值表示。一般来说,在连锁不平衡分析中, 野生种的 LD 值较低,而驯化种由于受到了正选择的作用,LD 值就会偏大。
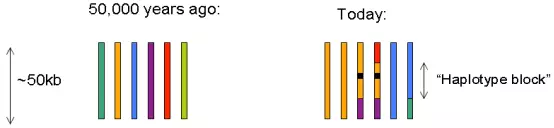 此处输入图片的描述
此处输入图片的描述
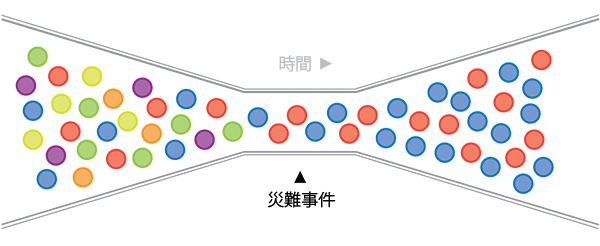
瓶颈效应:由于环境骤变(如火灾、地震、洪水等)或人类活动(如人工选择、驯化),使得某一生物种群的规模迅速减少,仅有一少部分个体能够顺利通过瓶颈事件,在之后的恢复期内产生大量后代。
 此处输入图片的描述
此处输入图片的描述
迁移压力(又叫基因流):由于某种原因,具有某一基因频率的群体的一部分移入基因频率与其不同的另一群体,并杂交定居,就会引起迁入群体的基因频率发生改变。
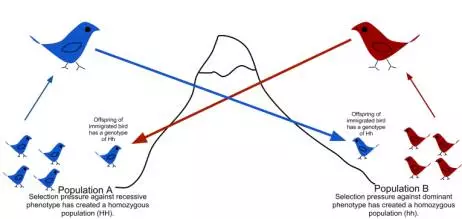 此处输入图片的描述
此处输入图片的描述


