系统发育基础知识介绍
系统发育学概念
 此处输入图片的描述
此处输入图片的描述
- 系统发生(或种系发生、系统发育,phylogeny)是指生物形成或进化的历史。系统发生学(phylogenetics)研究物种之间的进化关系,其基本思想是比较物种的特征,并认为特征相似的物种在遗传学上接近。系统发生研究的结果往往以系统发生树(phylogenetic tree)表示,用它描述物种之间的进化关系。
- 所有的生物都可以追溯到共同祖先,生物的产生和分化就像树一样生长、分叉。自然,我们以树的形式表现生物之间的进化关系。
- 系统发育树又称为系统进化树,是用一种类似树状分支的图形来概括各物种之间的亲缘关系,可用来描述物种之间的进化关系。
系统进化树介绍
 此处输入图片的描述
此处输入图片的描述
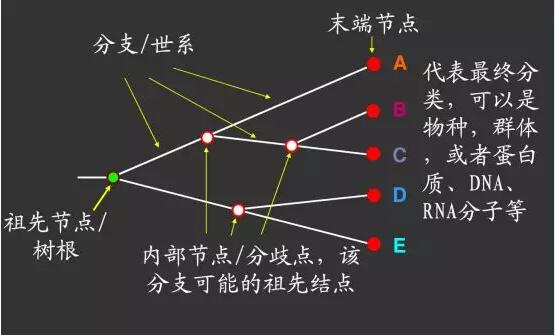
- 一般来说,系统发生树是一种二叉树。所谓树,实际上是一个无向非循环图。系统发生树由一系列节点(nodes)和分支(branches )组成,其中每个节点代表一个分类单元(物种或序列),而节点之间的连线代表物种之间的进化关系。
- 树的节点又分为外部节点(terminal node)和内部节点(internal node)。在一般情况下,外部节点代表实际观察到的分类单元,而内部节点又称为分支点,它代表了进化事件发生的位置,或代表分类单元进化历程中的祖先。
- 基于单个同源基因差异构建的系统发生树称为基因树(gene tree),这比称作物种树(species tree)更为合理。因为这种树代表的仅仅是单个基因的进化历史,而不是它所在物种的进化历史。物种树一般最好是通过综合多个基因数据的分析结果而产生。
系统发生树有许多形式:可能是有根树(rooted tree ,具有外群),也可能是无根树(unrooted tree,没有外群);
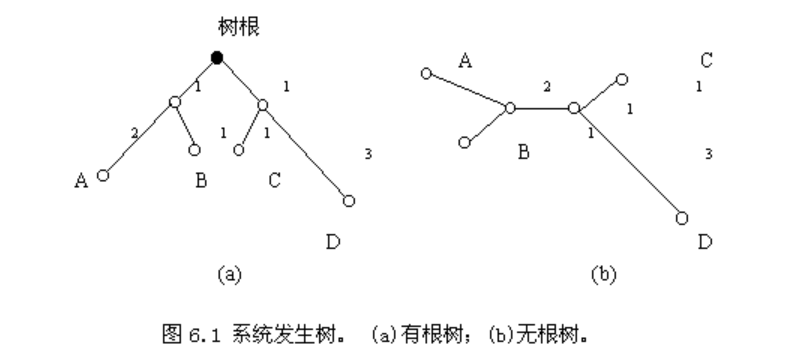
• 无根树:仅能表明分类单元之间分类关系,无法反应分化的先后
• 有根树:同时反映了分类单元间的分类关系和分化先后,因此可以用于分化时间的推断
• 有根树的根节点的选择:研究材料的全部分类单元的最近共同祖先
系统发生树性质
(1)如果是一棵有根树,则树根代表在进化历史上是最早的、并且与其它所有分类单元都有联系的分类单元;
(2)如果找不到可以作为树根的单元,则系统发生树是无根树;
(3)从根节点出发,到任何一个节点的路径均指明进化时间或者进化距离。
距离和特征
用于构建系统发生树的分子数据分成两类:(1)距离(distances)数据,常用距离矩阵描述,表示两个数据集之间所有两两差异;(2)特征(characters)数据,表示分子所具有的特征。
分子系统发生分析的目的是探讨物种之间的进化关系,其分析的对象往往是一组同源的序列。这些序列取自于不同生物基因组的共同位点。序列比对是进行同源分析的一种基本手段,是进行系统发生分析的基础,一般采用基于两两比对渐进的多重序列比对方法。通过序列的比对,可以分析序列之间的差异,计算序列之间的距离。
常用比对软件
系统发育树构建的第一步是进行多序列比对,常用的软件包括MEGA, cluster X,Muscle,phylip等。
| 软件 | 优点 | 缺点 |
|---|---|---|
| MEGA | 最常用的比对建树软件, 可视化图形界面,简单方便 | 比对速度慢,输出格式单一 |
| Clusterx | 可视化图形界面,可输出多种格式(如phy) | 比对速度较慢 |
| Muscle/phylip | 比对速度快 | 没有可视化界面,需要有一定编程基础去输入代码运用 |
系统发育树构建方法
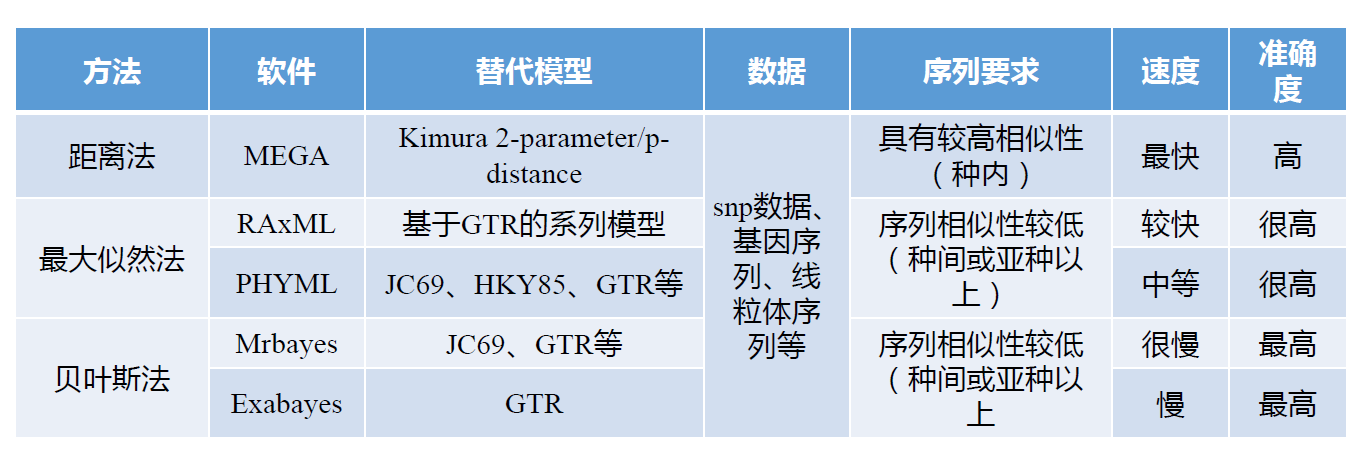
1、Distance-based methods 距离法:
(基于距离的方法:首先通过各个物种之间的比较,根据一定的假设(进化距离模型)推导得出分类群之间的进化距离,构建一个进化距离矩阵。进化树的构建则是基于这个矩阵中的进化距离关系。)
· Unweightedpair group method using arithmetic average(UPGMA)非加权分组平均法
· Minimum evolution(ME)最小进化法
· Neighbor joining(NJ)邻位归并法
2、Character-based methods 特征法:
(基于特征的方法:不计算序列间的距离,而是将序列中有差异的位点作为单独的特征,并根据这些特征来建树。)
· Maximum parsimony(MP) 最大简约法
· Maximum likelihood(ML) 最大似然法
其中非加权分组平均法已经较少使用。一般来讲,如果模型合适,最大似然法的效果较好。对近缘序列,有人喜欢最大简约法,因为用的假设最少。最大简约法,一般不用在远缘序列上,这时一般用邻位归并法或最大似然法.对相似度很低的序列,邻位归并法往往出现Long-branch attraction(LBA,长枝吸引现象),有时严重干扰进化树的构建。贝叶斯的方法则太慢。对于各种方法构建分子进化树的准确性,一篇综述(Hall BG. Mol Biol Evol 2005,22(3):792-802)认为贝叶斯的方法最好,其次是最大似然法,然后是MP。其实如果序列的相似性较高,各种方法都会得到不错的结果,模型间的差别也不大。不过现在文章普遍使用的最大似然法模型。
进化树评估
在实际应用中,我们需要评价一棵系统发生树的可靠性,这涉及两个问题,即整棵树和它的组成部分(分支)的置信度是多少?这样得到正确的树的可能性比随机选出一棵是正确的树的可能性大多少?
一种叫做自举法(bootstrapping)的有效的重采样技术已成为解决第一个问题的主要方法。自举检验(bootstrap test) 是一种重抽样技术,能粗略地量化这些置信度水平。造成统计误差的一个原因是数据采样误差,测量采样误差的一个好方法是,对于分析的对象多次采样,比较不同样本得到的估计值,估计值的分布可以说明一些问题。现在一般文章要求Bootstrap值1000。虽然根据严格的统计学概念,自展值要大于95%才较为可信,然而在实际应用中,特别是微生物等相似度比较大的分类中,一般大于50%就认为可信(小于50%隐去)。


